随着高通量测序、质谱等技术的发展和应用,目前发展出各种各样的微生物检测方法,其中16S rRNA扩增子测序和宏基因组测序是两种常用的方法。这两种方法都可以用于微生物多样性和功能的研究。
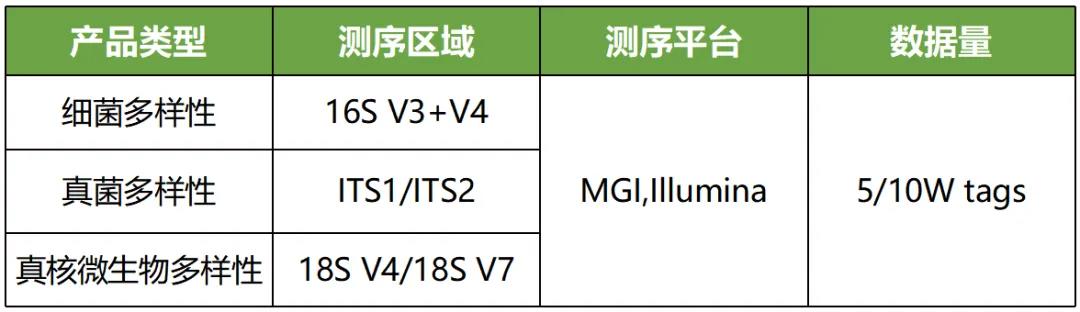
扩增子测序主要通过对特定长度的PCR产物进行测序分析,研究微生物多样性,群落组成差异以及物种间的进化关系。16S/18S/ITS扩增子测序分别针对细菌/真核生物/真菌的高变区进行PCR测序,基于合适的数据进行各分类水平的分类群注释;是分析微生物多样性的常用方法。扩增子测序方法以其低成本、短周期、丰富的分析内容,应用在各领域的微生物研究中。
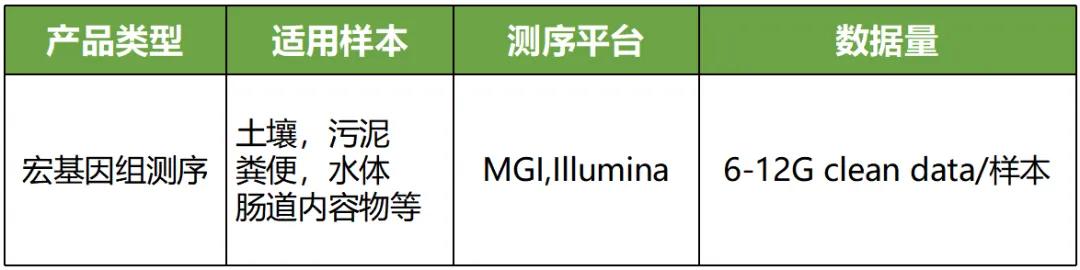
宏基因组测序是对样本中所有的微生物DNA进行高通量测序,经过序列处理分析,可以识别微生物群落的物种结构,进化关系,微生物群体的功能活性,探究致病或耐药性等分子机制,挖掘具有研究价值的分子通路等。
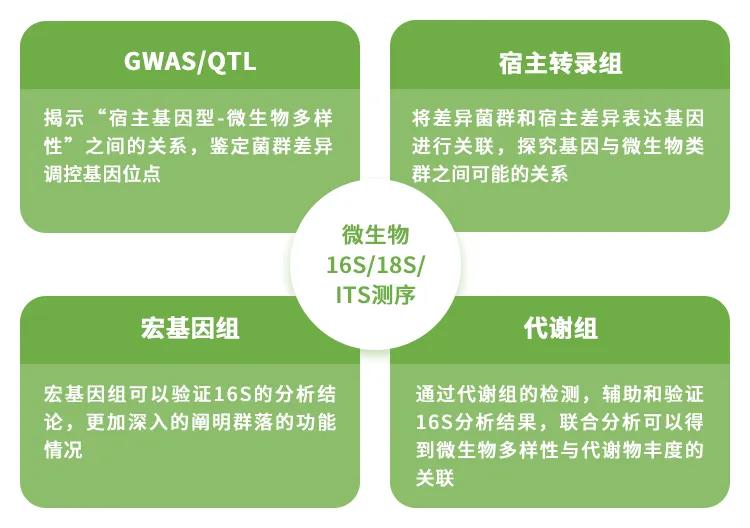
现阶段微生物研究转向更深入更广泛的复杂机制探讨,16S/18S/ITS扩增子与宏基因组测序、转录组、代谢组等其他组学结合,是最为频繁的研究方法;可以从多维度、多组学角度全面揭示环境变异和生物变异对微生物的影响。
微生物多样性研究策略
根据研究目的和菌群的差异,可以选择16S rRNA、18S rRNA、ITS等特定区域进行测序,解析微生物群落组成和丰度。
宏基因组研究策略
丁酸梭菌和碳水化合物活性酶有助于减少猪的脂肪沉积[1]
发表单位:浙江省农科院杨华,肖英平老师团队
发表时间:2024-01
发表杂志:iMeta(IF= 23.7)
技术路线:
研究框架:
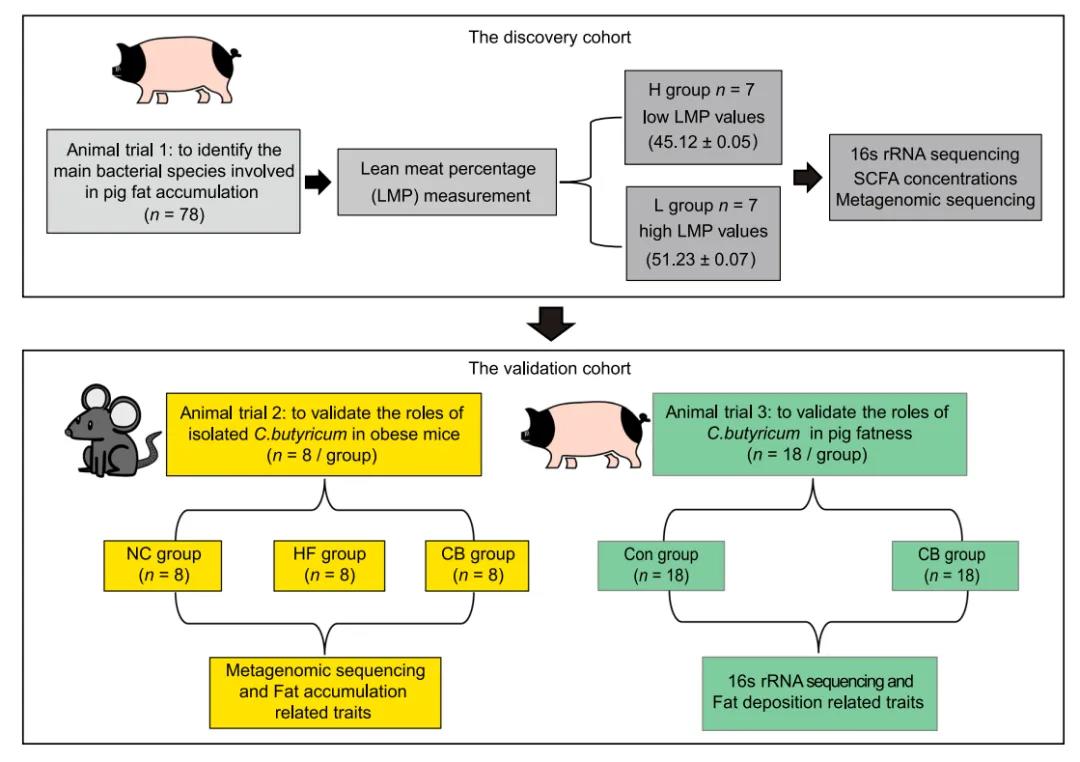
文章设计了三个动物试验:在第一个动物试验中,以78头雄性金华猪为研究对象,并进一步选择14头具有极端表型值的金华猪肠道内容物进行16S rRNA和宏基因组测序,以揭示不同脂肪沉积表型金华猪的微生物结构及功能差异。验证试验包括小鼠和猪的丁酸梭菌干预试验。对于小鼠试验,通过高脂饮食诱导小鼠肥胖模型,验证丁酸梭菌分离株对高脂饮食诱导的肥胖小鼠脂肪沉积和肠道微生物的作用。同时,进一步以金华猪为研究对象,通过丁酸梭菌饲喂试验,验证丁酸梭菌对猪体脂沉积的调控作用。
研究结论:
不同体脂表型金华猪不同肠段的微生物组成和功能存在显著差异;低体脂金华猪结肠中的短链脂肪酸含量较高,表明其碳水化合物发酵能力更强;丁酸梭菌是低脂肪沉积金华猪的代表性菌种,其基因组中碳水化合物活性酶GH13的比例较高;丁酸梭菌分离株可减缓肥胖小鼠的脂肪沉积并增加肠道微生物中GH13的丰度。
细尺度取样揭示了幼二穗短柄草根际微生物组与块状土壤的早期分化[2]
发表单位:美国加州大学Jillian F. Banfield和Romy Chakraborty团队
发表时间:2023-06
发表杂志:ISME(IF=11)
技术路线:
研究框架:
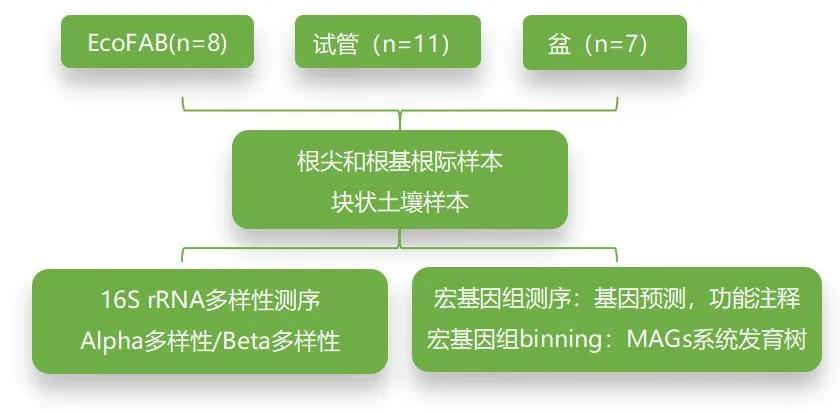
本文研究垂直空间上的,即根顶部,根基部和块状土壤的微生物差别。研究使用了3种不同的种植容器种植二穗短柄草:标准化制造的生态系统EcoFAB、试管、盆。从不同的容器中分别采集根基,根尖和块状土壤样本,基于16S rRNA测序和宏基因组测序分析对微生物群落进行研究分析,主要比较不同容器,不同取样部位的微生物的差异。
研究结论:
16S分析表明,根尖与根基部的微生物群落基本上相似,但与周围土壤中的微生物群落明显不同;标准化生长容器(EcoFAB)以及更传统的盆栽和试管对微生物群落的影响很小。宏基因组测序结果,预测基因KEGG注释后富集分析表明,与不同代谢途径和根部定殖相关的基因在根尖样品中高度富集,而与营养限制和环境压力相关的基因在块状土壤样品中富集。
参考文献
-
Ma, L., Tao, S., Song, T., Lyu, W., Li, Y., Wang, W., Shen, Q., Ni, Y., Zhu, J., Zhao, J., Yang, H., & Xiao, Y. (2024). Clostridium butyricum and carbohydrate active enzymes contribute to the reduced fat deposition in pigs. iMeta, 3(1), e160.
-
Acharya, S. M., Yee, M. O., Diamond, S., Andeer, P. F., Baig, N. F., Aladesanmi, O. T., Northen, T. R., Banfield, J. F., & Chakraborty, R. (2023). Fine scale sampling reveals early differentiation of rhizosphere microbiome from bulk soil in young Brachypodium plant roots. ISME Communications, 3(1), 54.
图文来源:北京康普森农业科技有限公司








 微信公众号
微信公众号
 下载app
下载app
 京公网安备 11010202008974号
京公网安备 11010202008974号