遗传变异通常分为两类:序列变异和结构变异。
基因组结构性变异(Structure Variantions,简称SVs)通常指基因组上大长度的序列变化和位置关系变化。基因组结构性变异类型很多,包括长度在50bp以上的长片段序列插入或者删除(Big Indel)、串联重复(Tandem repeate)、染色体倒位(Inversion)、染色体内部或染色体之间的序列易位(Translocation)、拷贝数变异(CNV)以及形式更为复杂的嵌合性变异[1]。
SV的类型主要有以下五种分别为:
插入 (Insertion, INS) :大于50bp的外源片段插入到染色体上。
缺失 (Deletion, DEL):染色体上一大段(>50bp)遗传物质被删除。
倒位(Inversions, INV):基因组中一段片段发生断裂后旋转180度,又插入到了相同的位置,那么结果就是基因组总长不变,但是这个片段首末端颠倒位置。
重复 (Duplication, DUP):是指基因组中一段序列被复制并且被插入到了基因组上,这样基因组上会有多个相同的长序列片段,这就导致了基因组发生了DUP结构变异。如果复制出来的序列插入到被复制序列的旁边那边我们称这段重复序列为串联重复序列(Tandem Duplication)。如果复制出来的序列没有紧邻被复制的序列,而且两段序列间有其他序列分隔,那么我们称这段重复序列为散在重复序列(Interspersed Duplication)。
易位(Translocation, TRA):基因组中一个片段断裂然后插入到了基因组中的其他区域,导致这个片段在基因组中的位置发生了变化。
图1.结构变异类型示意图
目前已知使用三种方法来检测SV:
1)基于阵列检测(array-base),包括微阵列基因组杂交(array CGH),但是这种方法只能检测某些SV类型,对小SV灵敏度较低。
2)基于短读长测序,即使用短读长测序获取有关SV的信息。但是由于在获取SV结构特征时有的算法具有一定局限性,比如比对或从头组装发生错误等。这也限制着此方法的准确性。
3)基于长度长测序,为了克服短读长测序的缺点,使用长度长来检测SV是近期比较流行的一种方法。但是改策略高成本低数据量限制了其广泛使用。
基于以上三种方法的优缺点,目前普遍使用的是第二种基于短读长的SV检测方法。因为短读长测序相对于长读长来说成本较低可以检测高深度规模样本(准确度不够,深度和数量来凑)。并且目前也有研究表明多种SV检测算法结合一起分析可以提高SV检出的灵敏度和准确性。
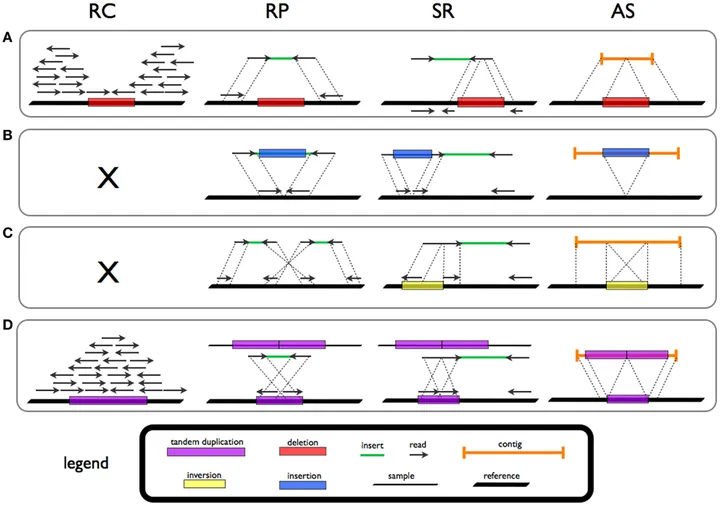
图2.SV 检测策略示意图:
A-缺失,B-插入, C-倒位和 D-串联重复;
四种分析策略:Read count (RC), Read-pair (RP),
Split-read (SR),和 de novo assembly(AS)[2]
所以本篇文章接下来主要介绍一下基于短读长测序检测SV的基本原理和简单的SV检测软件delly[3]的使用。
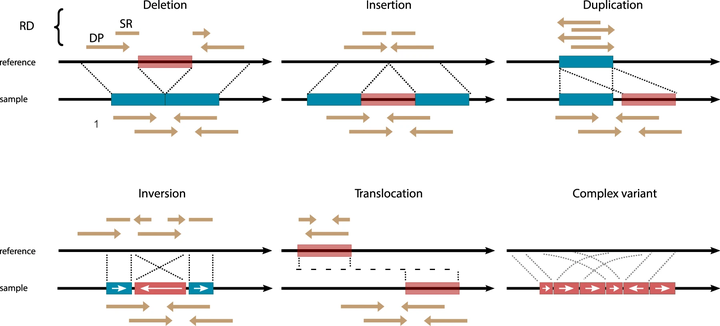
在使用短读测序结果分析SV之前,我们先明确一个概念就是:read深度(read depth,(RD)):参考基因组上某一碱基mapped reads个数。
在短读长检测SV的算法中,最核心的点就是根据SV的特殊结构找到SV的位置。而为了找到SV的特殊结构我们就要找到特殊的reads。一般而言普通reads pair mapped参考基因组上是一对方向相反但方向全部面向对方的一对reads。而找SV的结构就要找特殊mapped的reads pair,这里使用最多的就是不一致reads pair(Discordant pairs, DP)和分割read(Split reads, SR)。
RD或覆盖范围的变化大多表明存在较大的重复或删除,对于检测拷贝数变异 (CNV) 很有用。
DP是与预期在比对在基因组上相对距离或方向上不同的reads pair,DP最合适检测大型的SV,比如染色体易位或者倒位。
SR是同一个read 不同区域比对到参考基因组的距离较远的不同区域上。SR可以检测小片段的变异。
图3.不同结构变异的reads对齐模式
DELLY是非常经典的体细胞SV检测软件。它主要使用上述提到的方法来检测SV。具体使用步骤请参考github中DELLY官方教程(GitHub - dellytools/delly: DELLY2: Structural variant discovery by integrated paired-end and split-read analysis)。在此我们仅说明其中的简单使用。
Delly 需要为每个输入样本提供一个经过排序、索引和重复标记的 bam 文件。需要索引的参考基因组来识别拆分读段。SV 检出的常见工作流程概述如下:
Somatic变异检测
至少需要一个肿瘤样本和一个匹配的对照样本。
delly call -x hg19.excl -o t1.bcf -g hg19.fa tumor1.bam control1.bam
体细胞过滤需要制表符分隔的样本描述文件,其中第一列是样本 ID(如 VCF/BCF 文件),第二列是肿瘤或对照。
delly filter -f somatic -o t1.pre.bcf -s samples.tsv t1.bcf
在更大的对照样本组中对过滤的体细胞位点进行基因分型,以有效过滤假阳性和种系 SV。出于性能原因,可以对控制面板的每个样本并行运行。
delly call -g hg19.fa -v t1.pre.bcf -o geno.bcf -x hg19.excl tumor1.bam control1.bam ... controlN.bam
使用所有对照样品对体细胞SV进行后过滤。
delly filter -f somatic -o t1.somatic.bcf -s samples.tsv geno.bcf
Germline变异检测
delly call -g hg19.fa -o s1.bcf -x hg19.excl sample1.bam
将多个样本的合并到一起。
delly merge -o sites.bcf s1.bcf s2.bcf ... sN.bcf
在所有样本中对合并的 SV 位点列表进行基因分型。对于每个样本,这可以并行运行。
elly call -g hg19.fa -v sites.bcf -o s1.geno.bcf -x hg19.excl s1.bam
delly call -g hg19.fa -v sites.bcf -o sN.geno.bcf -x hg19.excl sN.bam
使用 bcftools merge 合并所有基因分型样本以获得单个 VCF/BCF。
bcftools merge -m id -O b -o merged.bcf s1.geno.bcf s2.geno.bcf ... sN.geno.bcf
对样本结果进行过滤整合(使用于至少20个样本)。
delly filter -f germline -o germline.bcf merged.bcf
参考文献
[1]Pellegrini M , Cole C , Schnhuth A ,et al.mini review detection of genomic structural variants from next-generation sequencing data[J]. 2018.DOI:10.3389/fbioe.2015.00092.
[2] Medvedev.Computational methods for the detection of structural variation in the human genome.[J]. 2012.
[3] DELLY: structural variant discovery by integrated paired-end and split-read analysis[J].Bioinformatics,2012,28(18):i333.DOI:10.1093/bioinformatics/bts378.
天津:18710280840/022-24986099
北京:400 1869 509
邮箱:marketing@kangpusen.com
地址:北京市昌平区中关村生命科学园生命园路4号院4号楼7层

图文来源:北京康普森农业科技有限公司













 下载app
下载app